- Presse
- Pressemeldungen 2021
- Wie Ethan-fressende Mikroben ihre Lieblingsspeise aufnehmen
Wie Ethan-fressende Mikroben ihre Lieblingsspeise aufnehmen
Die Forschungsergebnisse basieren auf der engen Zusammenarbeit mehrere Forschungsgruppen am Max-Planck-Institut für Marine Mikrobiologie in Bremen. So entdeckte ein Team um den Doktoranden Cedric Hahn und den Wissenschaftler Gunter Wegener erst kürzlich die Ethan-abbauenden Mikroben an heißen Quellen im Guaymas-Becken im Golf von Kalifornien in 2000 Metern Wassertiefe. Sie tauften ihre Entdeckung Ethanoperedens thermophilum, was soviel bedeutet wie „wärmeliebende Ethanfresser“.
Cedric Hahn aus der Forschungsgruppe "Molekulare Ökologie" gelang es im Anschluss, die ethanabbauenden Mikroben im Labor zu kultivieren. Gemeinsam mit zwei Kollegen aus der Forschungsgruppe „Mikrobielle Metabolismen“, Tristan Wagner und Olivier Lemaire, nahmen Hahn und Wegener diese Mikroorganismen genauer unter die Lupe. „Wir waren sehr erstaunt über das, was wir fanden. Zwar ähnelt das Enzym für Ethanabbau grundsätzlich seinem Gegenstück, dem Enzym für Methanabbau. In wichtigen Eigenschaften unterscheiden sich die beiden Enzyme aber grundlegend“, sagt Gunter Wegener, Wissenschaftler in der Forschungsgruppe "Tiefseeökologie und -technologie".

Ethanfresser sind auf das gleiche Enzym angewiesen wie Methanfresser
In Tiefseesedimenten trägt die geothermische Wärme zum Abbau von organischem Material zu Öl und natürlichen Gasen wie Ethan bei. Ethan wird nach aktuellem Forschungsstand von unterschiedlichen Mikroorganismen abgebaut, die ein so genanntes Konsortium bilden. Es handelt sich dabei um Archaeen, die das Gas abbauen, und Bakterien, die die dabei freigesetzten Elektronen mit Sulfat verbinden, welches im Meer reichlich vorhanden ist.
Bisher war es jedoch schwierig und langwierig, die biochemischen Prozesse der Konsortien im Labor zu untersuchen. Die Analysen beschränkten sich auf die methanabbauenden Mikroorganismen, die nur sehr langsam wachsen und sich nur alle paar Monate teilen. Die Entdeckung der Ethan-Mikroben hat Schwung in die Forschung gebracht, da sie deutlich schneller wachsen und sich wöchentlich verdoppeln. Dadurch steht schnell genug Biomasse für Analysen zur Verfügung und so konnten die Forschenden die Schlüsselenzyme, die die Oxidation von Erdgas katalysieren, erfolgreich aufreinigen und charakterisieren.
Um Ähnlichkeiten zwischen den Enzymen zu testen, die den Abbau von Ethan und Methan steuern, fügte Cedric Hahn seiner Kultur einen bekannten, molekularen Inhibitor der Reaktion mit Methan hinzu. Dieser stoppte auch die Ethan-Oxidation. „Das deutete darauf hin, dass die Ethan-oxidierenden Archaeen ebenfalls Ethan in ähnlichen Reaktionen wie beim Methanabbau und -aufbau aktivieren“, sagt Hahn. Solche Enzyme sind das Fachgebiet von Tristan Wagner, der sie seit vielen Jahren erforscht.
Mit erstaunlicher Präzision visualisiert
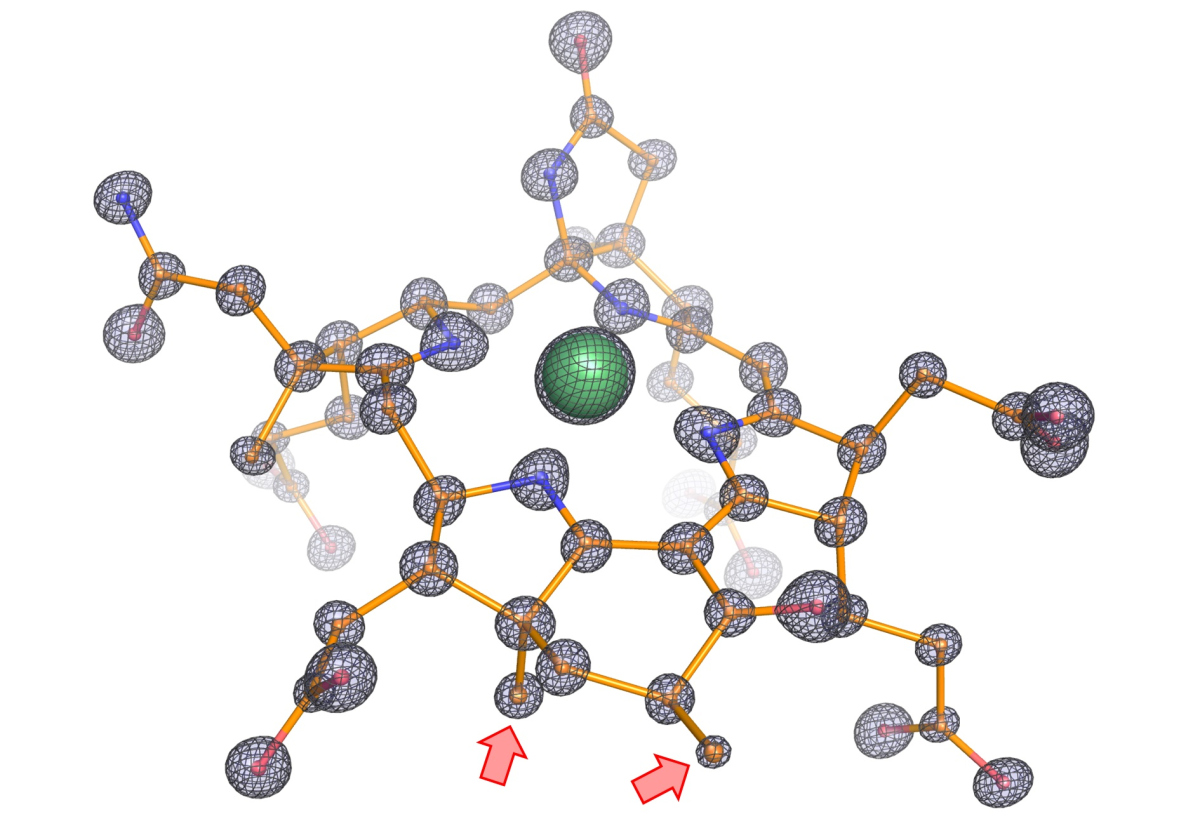
(©Max-Planck-Institut für Marine Mikrobiologie, O. Lemaire und T. Wagner)
Cedric Hahn und Olivier Lemaire, die beiden Erstautoren der jetzt in Science veröffentlichten Studie, versuchten dann, das für die Ethanfixierung verantwortliche Enzym aufzureinigen. „Das war eine große Herausforderung“, sagt Olivier Lemaire. „Normalerweise extrahieren wir die Enzyme aus viel größeren Mengen an Biomasse aus einer Kultur mit nur einem Mikroorganismus. Wir konnten aber schließlich eine ausreichende Menge an reinen Enzymen für Strukturanalysen gewinnen.“
Der nächste entscheidende Schritt war, Kristalle des Enzyms herzustellen, um seine dreidimensionale Struktur zu bestimmen. „Die Röntgenkristallographie liefert hervorragende Ergebnisse zu dieser Gruppe von Enzymen“, sagt Tristan Wagner, Leiter der Forschungsgruppe Mikrobielle Metabolismen und Experte auf dem Gebiet dieser Technik. „Wir analysierten die Kristalle mit gebeugter Röntgenstrahlung und konnten die Enzymstruktur in einer noch nie dagewesenen atomaren Auflösung zeigen. Das ermöglichte uns, sogar die Position einzelner Atome zu bestimmen und so ein äußerst präzises Bild der Struktur zu erhalten.“
Die atomare Auflösung zeigte den Forschenden unerwartete Fähigkeiten des Enzyms. „Uns fiel auf, dass die katalytische Kammer, in der die chemische Reaktion stattfindet, doppelt so groß ist wie in vergleichbaren Methan-Enzymen. Das macht durchaus Sinn, da Ethan größer ist als Methan“, sagt Lemaire. Der Katalysator der Reaktion, der Kofaktor, enthält außerdem zwei zusätzliche Methylgruppen. Diese Entdeckung wurde bestätigt von Jörg Kahnt vom Max-Planck-Institut für Terrestrische Mikrobiologie, einem weltweit anerkannten Experten dieses Kofaktors. „Wir haben ein Protein gefunden, das für das Bilden dieser Methylgruppen verantwortlich sein könnte, und es kommt nur bei Ethan-Konsumenten vor“, sagt Hahn. Da die Kammer größer ist, würde ein normaler Kofaktor nicht passen und so die Reaktion beeinträchtigen. Die Methylgruppen verankern ihn an der richtigen Stelle.
Außerdem enthält das Enzym einen Tunnel, der von außen zur katalytischen Kammer hinführt. Dieser Tunnel existiert in keinem der charakterisierten ähnlichen Enzyme. Möglich wurde der Nachweis dieses Tunnels durch die Zusammenarbeit mit Sylvain Engilberge am Paul Scherrer Institut in der Schweiz, wo die Proteinkristalle mit Xenon begast wurden. Das Xenon wurde in der Katalysatorkammer und dem Gastunnel nachgewiesen und somit seine Existenz belegt.
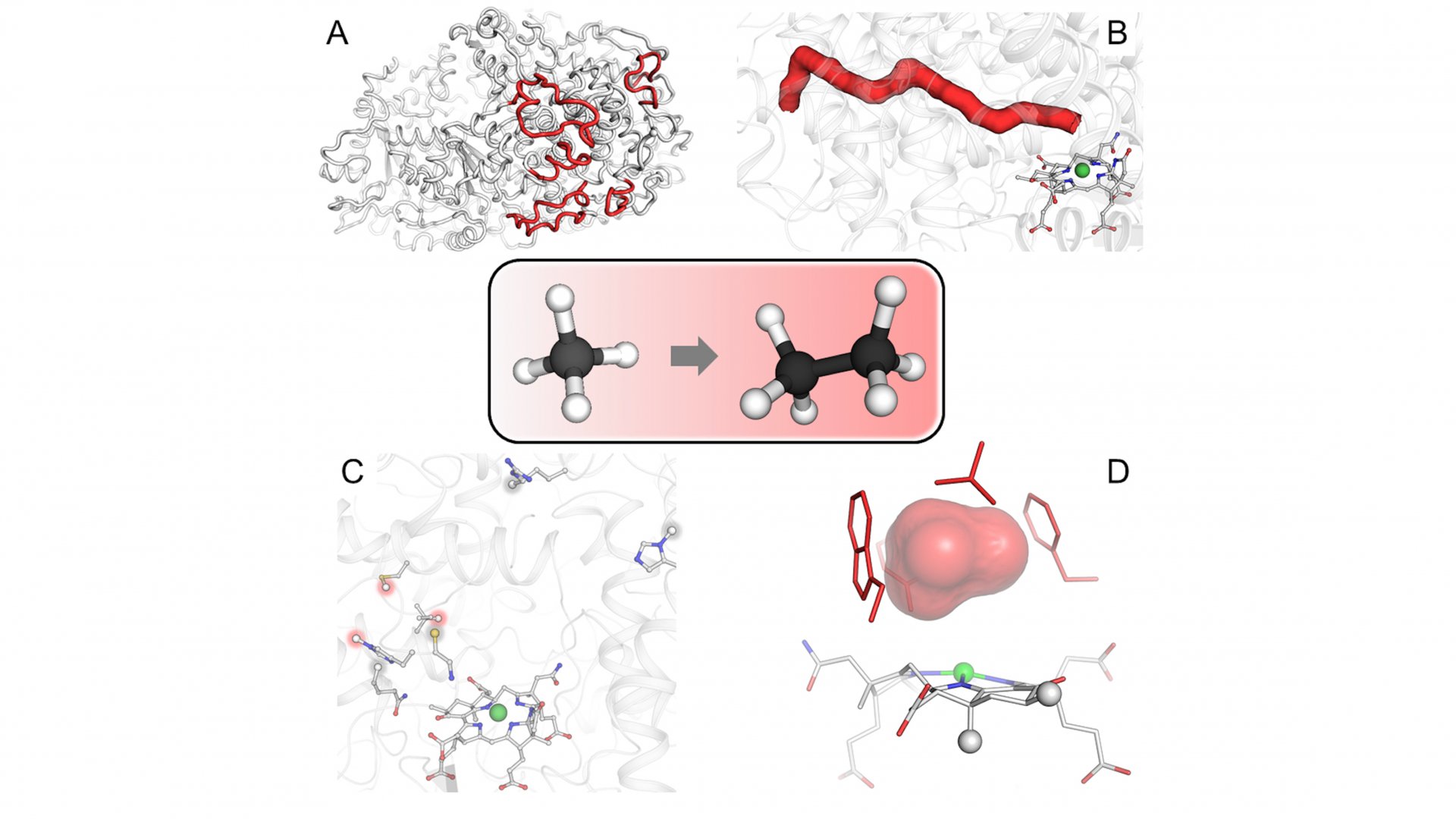
Nun rücken Propan und Butan in den Fokus
Die entdeckte Enzymstruktur verdeutlicht, wie sich diese Mikroben aus geothermisch aktiven Quellen auf die Ethanabscheidung spezialisiert haben. So ermöglicht diese Studie ein tieferes Verständnis des ersten Schritts des Ethanabbaus, der einzigen Energiequelle dieser Archaeen. „Unsere Erkenntnis, dass das für den Prozess verantwortliche Enzym spezifische Eigenschaften hat, um Ethan von anderen Alkanen zu unterscheiden, ist ein großer Schritt nach vorn. Zum Verständnis des gesamten Abbauprozesses ist es aber noch ein langer Weg“, sagt Tristan Wagner.
Wie geht es nun weiter? „Unsere bisherigen Arbeiten zeigen, dass die Aktivierung längerer Alkane ähnliche Enzyme erfordert“, sagt Gunter Wegener. „In einem nächsten Schritt wollen wir untersuchen, was die spezifischen Eigenschaften der Enzyme sein könnten, die den Abbau von Propan und Butan steuern.“
Originalveröffentlichung
Cedric J. Hahn*, Olivier N. Lemaire*, Jörg Kahnt, Sylvain Engilberge, Gunter Wegener, Tristan Wagner: Crystal structure of a key enzyme for anaerobic ethane activation, Science, July 2021
*Beide Autoren haben gleichberechtigt beigetragen
Beteiligte Institutionen
- Max-Planck-Institut für Marine Mikrobiologie, Bremen
- Max-Planck-Institut für Terrestrische Mikrobiologie, Marburg
- Paul-Scherrer-Institut (PSI), Villigen, Schweiz
- MARUM – Zentrum für Marine Umweltwissenschaften der Universität Bremen, Deutschland
Rückfragen bitte an:
Gruppenleiter
MPI für Marine Mikrobiologie
Celsiusstr. 1
D-28359 Bremen

Wissenschaftler
HGF MPG Brückengruppe für Tiefsee-Ökologie und -Technologie
MPI für Marine Mikrobiologie
Celsiusstr. 1
D-28359 Bremen
|
Raum: |
1335 |
|
Telefon: |
