- Forschung & Instrumente
- Wie wir forschen - Unsere Instrumente und Methoden
- Instrumente und Methoden
- An Land
- MALDI-Imaging Massenspektrometer
MALDI-Imaging Massenspektrometer

Was ist ein MALDI-Imaging Massenspektrometer?
Bei MALDI-Imaging Massenspektrometrie handelt es sich um eine bildgebende Methode zur Analyse chemischer Verbindungen und deren räumlicher Verteilung in einer Probe. Die Abkürzung MALDI bedeutet übersetzt Matrix-unterstützte Laser-Desorption/Ionisation. Man kann damit erkennen, an welcher Stelle sich bestimmte Verbindungen in einer Probe befinden. So ist nicht nur sichtbar, welche Zucker und/oder Fette beispielsweise in einer Muschel enthalten sind, sondern auch, wo sie in der Muschel vorhanden sind. Wissenschaftlerinnen und Wissenschaftler können mit diesen Informationen Rückschlüsse darauf ziehen, wie der Stoffwechsel der Muschel funktioniert.
Der Vorteil gegenüber einem Massenspektrometer verbunden mit einem Gas- oder Flüssigchromatographen besteht in dieser zusätzlichen örtlichen Information. Um diese chemischen Bilder zu erzeugen scannt ein Laser der MALDI-Quelle die biologische Probe in einem Raster von bis zu zehn Mikrometer – also zehnmal dünner als ein menschliches Haar. So können sehr feine Details und Strukturen, wie zum Beispiel einzelne Tierzellen, sichtbar gemacht werden. Da jeder Rasterpunkt circa eine Sekunde Messzeit benötigt, können Messungen von weniger Quadratmillimetern bei hoher Auflösung mehrere Tage dauern. Das bedeutet, dass nicht die ganze Muschel gemessen werden kann, sondern nur Teile, zum Beispiel die Kiemen.
Wie funktioniert das MALDI-Imaging Massenspektrometer?
Zunächst wird das biologische Material für die Messung so vorbereitet, dass sowohl die Gewebestruktur, als auch die räumliche Verteilung der chemischen Verbindungen im Gewebe intakt bleibt. Dazu wird mit flüssigem Stickstoff schockgefrorenes Gewebe in ein Trägermaterial eingebettet. Anschließend wird es gefroren in einem Cryotom bei -25 Grad Celsius in zehn Mikrometer dünne Schnitte geschnitten und glatt auf einem Objektträger aufgebracht. Um die Struktur des Gewebeschnittes festzuhalten, wird ein Foto mit einem Licht-Mikroskop gemacht. Anschließend wird mit einem Airbrush-artigem Sprayer eine Matrixlösung aufgebracht. Die Matrix dringt in die oberste Schicht des Gewebes ein, kristallisiert beim Trocknen und schließt die chemischen Verbindungn (wie Zucker, Eiweiß oder Fettsäuren) in feine Kristalle ein. Dabei gilt: Je feiner die Kristalle, desto genauer im Anschluss das Messergebnis.
Während der Messung schießt ein fokussierter Laser auf die Gewebeprobe und rastert eine bestimmte Fläche (zum Beispiel 100 x 100 Mikrometer) mit einer Rasterweite von zehn Mikrometern ab. Das heißt, der Laser schießt alle zehn Mikrometer auf das Gewebe. Mit jedem Schuss werden die Analyten herausgelöst (Desoption) und erhalten in der MALDI-Quelle eine positive Ladung (Ionisation), die von den Matrixmolekülen übertragen wird. Die geladenen Ionen werden in der Orbitrap, einem Ionenfallen-Massendetektor, nach ihrem Masse-zu-Ladung-Verhältnis sortiert und detektiert. Das Ergebnis des gemessenen Areals wird in einem Bild zusammengefasst, welches in 10x10 Mikrometer große Pixel unterteilt ist. Jedes dieser Pixel enthält in einem Spektrum die Information, welche verschiedenen Masse-zu-Ladung-Verhältnisse an dieser Stelle gemessen wurden. Mit dem Orbitrap-Massenspektrometer kann dieses Verhältnis sehr genau gemessen werden, bis vier Stellen nach dem Komma. Diese hohe Genauigkeit nennt man hochauflösende Massenspektrometrie. Man erhält so zu jedem Pixel ein Spektrum, das Rückschlüsse auf die elementare Zusammensetzung der chemischen Verbindungen zulässt und wichtige Informationen für die Strukturaufklärung liefert.
Sowohl die gemessenen Spektren als auch das dazugehörige Bild der örtlichen Verteilung kann man zusätzlich in Datenbanken laden. Dort erhält man noch weitere Informationen zu den Verbindungen und deren Vorkommen in verschiedenen Gewebetypen.


Das MALDI-Imaging Massenspektrometer im Einsatz
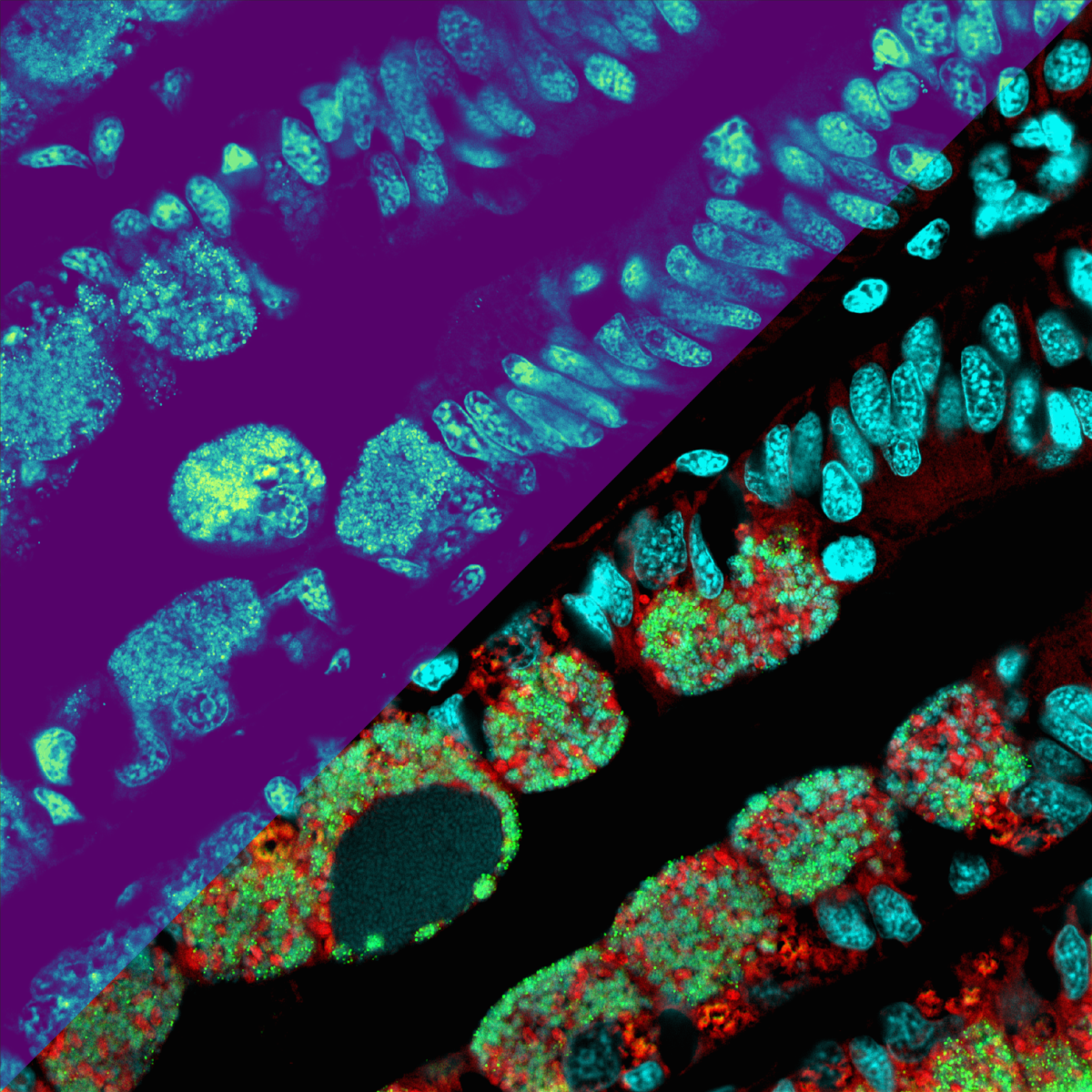
Schnappschuss von Molekülen in einer Tiefsee-Symbiose
Ein Team von Forschenden um Benedikt Geier und Manuel Liebeke vom Bremer Max-Planck-Institut für Marine Mikrobiologie haben eine Methode entwickelt, mit der man einzelne Bakterien identifizieren und gleichzeitig erkennen kann, welche Zwischenprodukte des Stoffwechsels in der Zelle vorhanden sind. Mit der neuen Methode erforschen sie, wie Bakterien als symbiotische Untermieter in Tiefseemuscheln wohnen und überleben. Liebeke und sein Team analysierten Hunderte von Stoffwechselprodukten auf einer Fläche kleiner als ein Quadratmillimeter und nutzen dafür das MALDI-Massenspektrometer. So können sie erkennen, wie symbiotische Mikroben in ihrem Wirt leben und auf chemischer Ebene interagieren. Sie machen quasi einen Schnappschuss der Bakterien bei der Arbeit – so, wie sie in ihrer natürlichen Umwelt, in diesem Fall innerhalb einer tierischen Zelle, aktiv sind.
Korrekte Schlussfolgerungen aus den Bildern sind aber nur möglich, wenn auch bekannt ist, wer sie erzeugt oder nutzt. Um dieses Problem zu lösen, nutzten die Forschenden zusätzlich eine weitere Methode, die sogenannte Fluoreszenz in situ Hybridisierung oder kurz FISH, um einzelne Bakterienzellen in der jeweiligen Probe zu identifizieren. Durch die Kombination mit FISH können sie MALDI- Bilder nun sinnvoll erklären und den Bakterien im Muschelgewebe zuordnen.
Mehr Informationen dazu gibt es in der Pressemitteilung „Das geheime Leben der Mikroben: Schnappschuss von Molekülen in einer Tiefseesymbiose“
Artikel in der Fachzeitschrift Biospektrum: "Die chemische Sprache von Symbiosen sichtbar machen"
Für diese innovative Methode erhielt Benedikt Geier den MSI Award, der alljährlich für herausragende wissenschaftliche Arbeiten mithilfe bildgebender Verfahren mit einem Massenspektrometer verliehen wird. Lesen Sie mehr dazu hier.
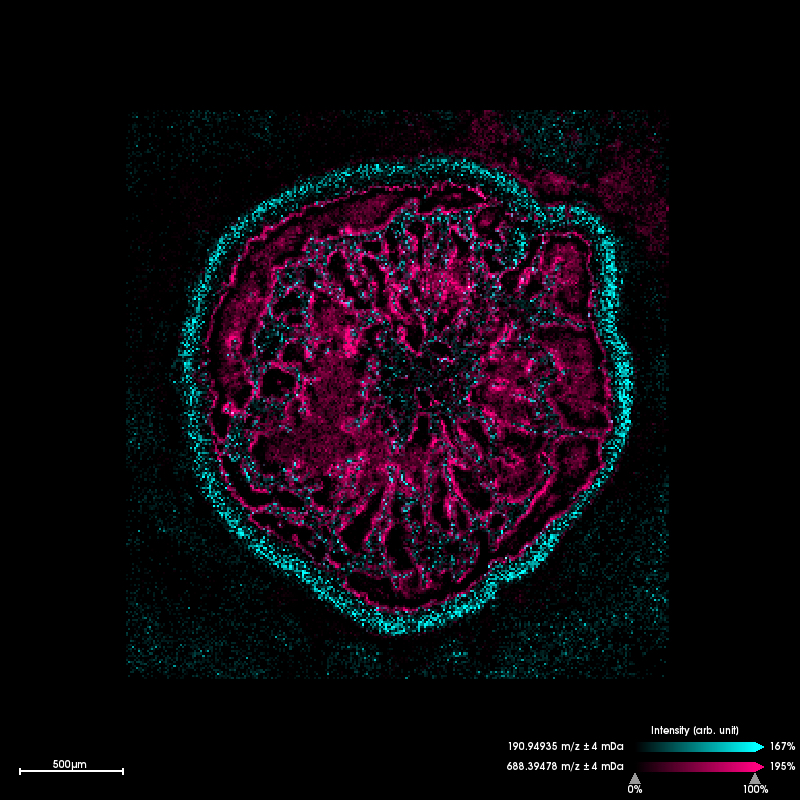
Seegraswurzeln
Das Erzeugen von Bildern für einzelne chemische Verbindungen klappt nicht nur mit tierischem Gewebe sondern funktioniert auch mit Pflanzen. Ein Beispiel aus unserer Forschung sind Seegräser. Hier dargestellt ist der Querschnitt durch eine Seegraswurzel und wie dort bestimmte chemische Verbindungen verteilt sind. Zum einen sieht man Stoffe die nur außen um die Wurzel vorkommen und damit direkt mit dem Sediment interagieren, andere Verbindungen kommen nur im Inneren der Wurzel vor und man kann sich vorstellen das dies zum Beispiel Speicherstoffe wie Lipide sind.
Wer nutzt das MALDI-Imaging Massenspektrometer?
Das MALDI-Imaging Massenspektrometer wird zumeist von der Gruppe Metabolische Interaktionen der Abteilung Symbiose verwendet. Aber auch andere Forschende des Instituts und GastwissenschaftlerInnnen haben die Möglichkeit, das Gerät bei Bedarf zu nutzen. Denn durch eine fachübergreifende Zusammenarbeit mehrerer Arbeitsgruppen ergeben sich spannende Projekte, von denen alle Beteiligten lernen können.
Kontakt
Gruppenleiter
MPI für Marine Mikrobiologie
Celsiusstr. 1
D-28359 Bremen
|
Raum: |
3244 |
|
Telefon: |

Technische Angestellte
MPI für Marine Mikrobiologie
Celsiusstr. 1
D-28359 Bremen
|
Raum: |
2506 |
|
Telefon: |
