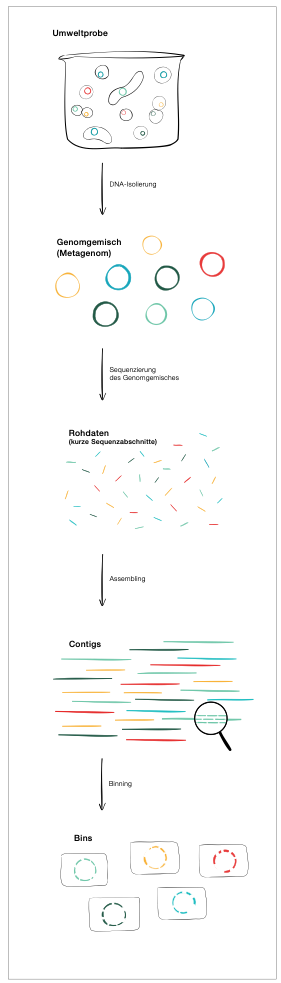Metagenomik
Was ist Metagenomik?
Stell dir vor, vor dir liegt ein Haufen schöner Bücher: Leider wurden sie in kleine Schnipsel zerschnitten, voller einzelner Wörter und Satzfragmente. Und deine Aufgabe ist es nun, Ordnung in dieses Chaos zu bringen. Am besten suchst du erstmal nach Schlüsselwörtern. Steht "Faust" auf dem Schnipsel, gehört er vermutlich zum gleichnamigen Werk von Goethe. Und wenn "Neusprech" oder "Big Brother" vorkommt, hast du es bestimmt mit 1984 von George Orwell zu tun. "Muggel" und "Hogwarts" wiederum weist eindeutig auf Bücher aus der Harry Potter-Reihe. So kannst du dich Stück für Stück durch den Papierhaufen wühlen und versuchen, die Schnipsel zu ordnen und zusammenzusetzen. Wurden mehrere Exemplare des gleichen Buchs unterschiedlich zerschnitten, erleichtert das die Sache – durch überlappende Textfragmente erhältst du zusätzliche Hinweise auf die richtige Zusammensetzung der Texte.
Metagenomik funktioniert gar nicht so anders. Statt der Bücher haben die Forschenden es dabei mit den Genomen von Mikroorganismen zu tun, die sie zerschneiden und wieder zusammensetzen. Die Papierschnipsel sind Fragmente des Genoms, sogenannte Sequenzabschnitte. Beim Zusammenfügen der Einzelteile hilft zum Glück ein Computer. Es gibt mittlerweile sehr gute Algorithmen, um dieses Puzzle zu lösen. Manche Sequenzabschnitte sind auch schon bekannt und in Datenbanken verzeichnet und können helfen, bestimmte Sequenzen zu Organismengruppen oder Funktionen zuzuordnen.
Mithilfe der Metagenomik wird es so möglich, eine Umweltprobe – also einem Gemisch verschiedenster Organismen, wie sie in der Umwelt zusammenleben – zu analysieren und zu erfahren, welche Organismen darin leben und welche Gene und damit Stoffwechselwege, Interaktionen oder Verteidigungsstrategien sie beherrschen. Das unterscheidet sie von klassischen mikrobiologischen Methoden, bei denen gezielt nach einzelnen Organismen(gruppen), deren Genomsequenzen bekannt sind, gesucht wird. Die Methode, mit der wir am Computer anschließend Ordnung in die metagenomischen Analysen und Daten bringen, nennt man Bioinformatik.
Metagenomische Methoden erlauben uns also zudem, Mikroorganismen unabhängig davon, ob sie im Labor kultiviert werden können, zu identifizieren damit auch die Dynamiken in den Populationen über Zeiträume von Tagen bis Jahren zu untersuchen. So erlangen wir einen Einblick in die Komplexität und den Wandel der Gemeinschaften der kleinsten Bewohner unseres Planeten. Allerdings kann die Metagenomik oft nicht allein stehen – ohne die umfassende Arbeit der klassischen Mikrobiologie könnten wir ihre Ergebnisse nicht interpretieren.
Wie funktioniert Metagenomik?
Wie funktioniert Metagenomik?
In der Umweltprobe finden wir ein Gemisch der Genome verschiedenster Organismen, das Metagenom. Für die effiziente Sequenzierung etwa des Metagenoms einer Bakteriengemeinschaft müssen wir die einzelnen Genome zunächst in unzählige Bruchstücke zerlegen. Die Sequenzierung liefert uns daraus zahlreiche ungeordnete Sequenzabschnitte, je nach benutzter Technologie mit einer Länge von etwa 100 bis hunderttausenden Basen.
Trotz enormer Fortschritte bei den Leselängen sind die Teilstücke viel kürzer als die typischen Genome der Mikroorganismen. Daher muss man die Stücke zu größeren Fragmenten zusammenpuzzeln, den sogenannten Contigs. Contigs sind zusammenhängende Sätze überlappender DNA-Stücke, die aus derselben genetischen Quelle – in unserem Fall der gleichen Bakterienart – stammen. Diese sind einige Kilobasen bis etwa eine Megabase lang und damit lang genug, um sie sinnvoll sortieren zu können.
Die Contigs können wir in feiner weiterer Puzzlearbeit so in Gruppen zusammenfassen, dass daraus wiederum mehr oder weniger vollständige Genome einzelner Bakterienarten entstehen. Dieses Verfahren nennen wir Binning. Dabei werden die Contigs in Abhängigkeit ihrer Basensequenz und Häufigkeit in "Schachteln", die sogenannten Bins, gruppiert. Ein Bin ist eine Schachtel, in die wir Teile des großen Metagenom-Puzzles sortieren, die mit hoher Wahrscheinlichkeit von einer Bakterienart stammen. Bei guter Vollständigkeit bezeichnet man diese Bins als aus "Metagenomen assemblierte Genome", kurz MAGs.
Die Grafik visualisiert das Prinzip.
Metagenomik im Einsatz
Mithilfe der Metagenomik untersuchen wir beispielsweise den Abbau von Algenblüten in der Nordsee: Jedes Jahr im Frühling, wenn die Tage länger werden und die Sonneneinstrahlung zunimmt, vermehren sich viele Phytoplanktonarten sehr schnell, ihre Population wächst exponentiell. Diese Die Algenblüten sind für den Kohlenstoffkreislauf von globaler Bedeutung. Im Zuge der Photosynthese wandeln die kleinen Algen Kohlendioxid in Zuckerverbindungen und Sauerstoff um. Innerhalb weniger Wochen gehen den Algen die Nährstoffe aus und Fressfeinde machen sich breit, die Algenblüte bricht zusammen. Bakterien vermehren sich massenhaft, sie bauen die übrig gebliebene Algenbiomasse ab, der gebundene Kohlenstoff wird dabei größtenteils wieder frei. Diese Vorgänge sind bis heute nicht genau verstanden. Welche Meeresbakterien wachsen wann? Wie verarbeiten sie die Algenbiomasse und setzen den Kohlenstoff wieder frei? Welche Prozesse stecken dahinter?
Dies ist nur ein Anwedungsbeispiel von vielen. Die Metagenomik wird in unserem Institut sehr oft verwendet. Es folgen ein paar konkrete Projektbeispiele aus verschiedenen Gruppen.
Projektbeispiele
Einfacher als erwartet: Die bakteriellen Aufräumarbeiten nach Algenblüten sind gar nicht so komplex
Algenblüten sorgen regelmäßig für schöne, verwirbelte Satellitenbilder von Seen und Ozeanen. Ab und an hört man auch von Ansammlungen giftiger Algen, die Fische, Menschen und andere Tiere gefährden. Weniger bekannt ist, was für eine bedeutende Rolle sie im globalen Kohlenstoffkreislauf spielen. Eine aktuelle Studie liefert nun überraschende Fakten über den Kohlenstofffluss in Algenblüten. Unerwartet wenige Bakteriengruppen mit einer begrenzten Anzahl von Genen zersetzen den größten Teil der vorhandenen Algenzucker.
Zur Pressemeldung https://www.mpi-bremen.de/Einfacher-als-erwartet.html
Verschollen auf hoher See: Fern der Küste lebt Thioglobus perditus aus ihrem Vorratspack
Der Bakterienstamm SUP05 gibt Forschern einige Rätsel auf. Wieso etwa finden sich diese Mikroben im offenen Ozean, obwohl es für sie dort keine Lebensgrundlage gibt? Denn SUP05-Bakterien nutzen zur Energiegewinnung die Schwefelverbindung Sulfid, und das findet sich meist nur nahe den Küsten. Eine Gruppe Forschender vom Max-Planck-Institut für Marine Mikrobiologie in Bremen hat nun in Zusammenarbeit mit dem GEOMAR und der Universität Kiel einige Antworten gefunden: Auf einer Ausfahrt mit dem Forschungsschiff Meteor entdeckten die Forscher im Meer vor Peru einen Vertreter dieses Bakterienstammes, der seinen eigenen Vorrat an Schwefel mit sich trägt. Außerdem gelang es den Forschenden, das komplette Genom des Bakteriums zu entschlüsseln. Die Mikrobe hat quasi einen Personalausweis ausgestellt bekommen. Name: Thioglobus perditus, sinngemäß die auf See „verschollene Schwefelkugel“. Anhand des Genoms entwickelten siedann eine Gensonde, mit deren Hilfe sie diese Mikrobe nun jederzeit genau identifizieren können.
Zur Pressemeldung https://www.mpi-bremen.de/Verschollen-auf-hoher-See-Fern-der-Kueste-lebt-Thioglobus-perditus-aus-ihrem-Vorratspack.html
Viele Köche verderben nicht den Brei: Hohe Symbiontenvielfalt wappnet den Wirt für alle Fälle
Tiefseemuscheln, die sich mit Hilfe bakterieller Symbionten ernähren, beherbergen überraschend viele Untermieter: Bis zu 16 verschiedene Bakterienstämme wohnen in den Kiemen der Muschel, jeder mit eigenen Fähigkeiten und Stärken. Dank dieser Vielfalt an symbiotischen Partnerbakterien ist die Muschel für alle Eventualitäten gewappnet.
Zur Pressemeldung https://www.mpi-bremen.de/Viele-Koeche-verderben-nicht-den-Brei-Hohe-Symbiontenvielfalt-wappnet-den-Wirt-fuer-alle-Faelle.html