Durchfluss-Zytometrie
Was ist Durchfluss-Zytometrie?
Durchflusszytometrie erlaubt das Messen verschiedener Eigenschaften einzelner Zellen in einem Wasserstrahl. Am Max-Planck-Institut für Marine Mikrobiologie nutzen wir die Durchflusszytometrie einerseits, um die Anzahl von Zellen in Seewasserproben und in Zellkulturen festzustellen. Andererseits können wir mit der Technik Zellen mit hoher Geschwindigkeit und in hoher Reinheit gemäß ihrer Eigenschaften sortieren, um sie im Anschluss weiter molekularbiologisch zu analysieren.
Für die molekularbiologische Forschung stellt die Durchflusszytometrie eine wichtige Ergänzung dar. Die Möglichkeit, Tausende von Zellen pro Sekunde zu analysieren und gleichzeitig multiple Parameter wie die Fluoreszenz von Photosynthesepigmenten oder spezifischen Fluoreszenzfarbstoffen aufzunehmen, macht die Durchflusszytometrie zu einem Standardwerkzeug in der Planktonforschung. Zusätzlich lässt das durchflusszytometrische Sortieren eine physikalische Trennung und kultivierungsunabhängige Anreicherung von wohldefinierten Zellpopulationen zu.
Die Durchflusszytometrie ist am Max-Planck-Institut für Marine Mikrobiologie eine eigene Arbeitsgruppe. Dort verbessern wir ständig unsere durchflusszytometrischen Methoden und verknüpfen sie mit neuen molekularbiologischen Ansätzen, um tiefere Einblicke in die jeweilige ökologische Rolle von Mikroorganismen in verschiedenen marinen Habitaten zu gewinnen.
Wie funktioniert die Durchfluss-Zytometrie?
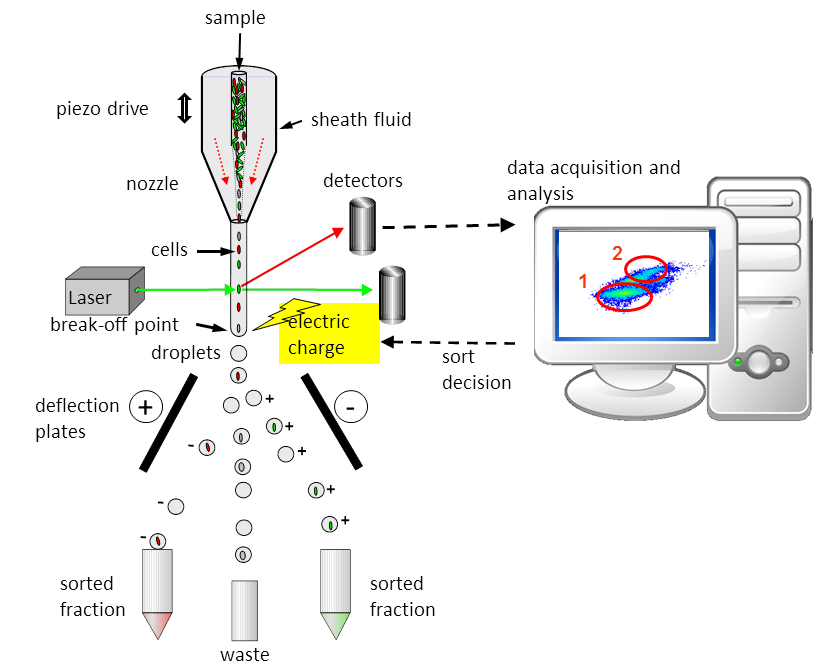
Bei der Durchflusszytometrie wird die Probe mit den Zellen in die Mitte eines kontinuierlich fließenden Wasserstrahls (auch Hüllflüssigkeit genannt) injiziert. Im weiteren Verlauf wird der Wasserstrahl mittels einer Düse auf einen Durchmesser von weniger als 1/10 Millimeter verjüngt. Die eingeschlossenen Zellen werden dadurch in die Mitte des Wasserstrahls fokussiert und verlassen die Düse wie Perlen auf einer Schnur. Die exakt ausgerichteten Zellen passieren den Laserstrahl einzeln im Beobachtungspunkt und können so vom Laserlicht angeregt werden. Je nach Größe und Färbung der Zelle werden die Lichtsignale der Zellen durch Linsen (nicht abgebildet) auf die Detektoren fokussiert, dort in elektrische Signale umgewandelt und weiter in eine Software am Rechner eingespielt.
Im Sortiermodus können die Zellen zudem nach bestimmten Eigenschaften getrennt werden. Dafür legen die Forschenden Bedingungen fest, die die Zellen erfüllen müssen. Die Düse schwingt in hochfrequenten Schwingungen, was dazu führt, dass der Wasserstrahl unterhalb des Beobachtungspunktes zu definierten Tröpfchen abreißt. Falls eine Zelle detektiert wird, die die vorgegebenen Parameter erfüllt, wird die entsprechende Zelle mit ihrem Tröpfchen elektrisch geladen. Die geladenen Tröpfchen mit den Zellen werden gemäß ihrer Ladung (positiv oder negativ) abgelenkt und landen in einem Reaktionsgefäß. Abgebildet ist hier ein Zwei-Wege-Sorter, der zwei Populationen gleichzeitig sortieren kann.
Wir haben am Institut aktuell drei verschiedene Durchflusszytometer. Sie unterscheiden sich unter anderem in Größe, Sensibilität und Ausstattung. Alle drei können transportiert werden und sind für den Einsatz an Bord von Forschungsschiffen geeignet. Weitere Informationen gibt es auf der Abteilungsseite der Forschungsgruppe Durchflusszytometrie.
Die Durchfluss-Zytometrie im Einsatz
Analyse unbekannter Mikroben vereinfacht
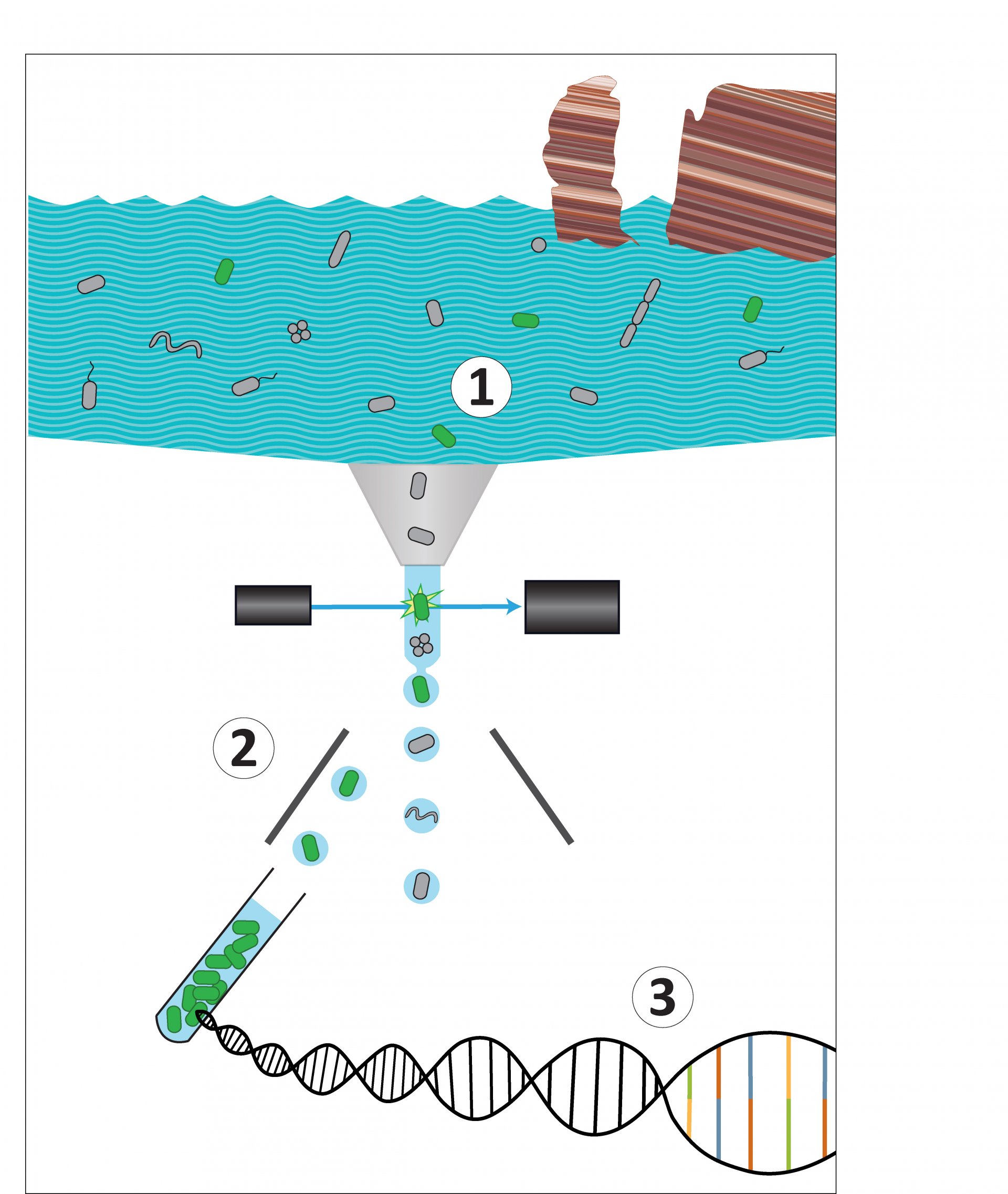
Marine Mikroben mögen es, verstecken zu spielen. So tauchen manche Bakterien immer wieder in Proben auf, wollen aber im Labor einfach nicht wachsen. Gleichzeitig sind es in den Umweltproben jeweils zu wenige, um ihre Identität über eine Gen-Analyse zu enthüllen.
Die Lösungs-Idee: Mit einem Durchflusszytometer die Zellen der interessierenden Spezies vor dem Sequenzieren der DNA aussortieren. So wird die Diversität geringer und die eigentlich nicht so häufige Spezies kann sich nicht mehr so gut verstecken. Die Wissenschaftlerinnen und Wissenschaftler markierten also mit der FISH-Methode DNA-Abschnitte der Mikroben mit fluoreszierenden Farbstoffen. So können sie die Bakterien erkennen und aussortieren. Allerdings gibt es dabei zwei Herausforderungen: Erstens muss das Farbsignal sehr stark sein, damit die Zellen während des Sortierens gut zu finden sind. Zweitens darf aber die DNA durch den Farbstoff nicht beeinträchtigt werden, da sie für eine qualitativ hochwertige Genom-Analyse im Anschluss möglichst intakt sein muss.
Gemeinsam mit Forschenden des Joint Genome Institute testete ein Team um Anissa Grieb, wie dieser Balanceakt gelingen kann. Erfolg hatten sie am Ende mit einer kürzlich entwickelten Variante der FISH-Methode, und zwar mit der Hybridisierungs-Kettenreaktion (HCR-FISH), die sie durch Anpassung verschiedener Parameter wie der Temperatur für ihre Zwecke optimierten. Zunächst haben sie diese Vorgehensweise mit Reinkulturen im Labor optimiert und anschließend erfolgreich mit den Umweltproben getestet, die vor Helgoland genommen wurden.
- Mehr Informationen dazu gibt es in der Pressemitteilung "Analyse unbekannter Mikroben wird einfacher"
- Das Paper zum Thema ist hier erhältlich: Grieb A, Bower RM, Oggerin M, Goudeau D, Lee J, Malmstrom RR, Woyke T, Fuchs BM. 2020. A pipeline for targeted metagenomics of environmental bacteria. Microbiome 8:21 https://doi.org/10.1186/s40168-020-0790-7
Wer nutzt die Durchfluss-Zytometrie?
Wissenschaftlerinnen und Wissenschaftler der Abteilung Molekulare Ökologie. Sie steht aber auch anderen Forschenden des Instituts offen sowie externen Forschenden im Rahmen von Kooperationsprojekten.
Kontakt
Gruppenleiter
Forschungsgruppe Durchflusszytometrie
MPI für Marine Mikrobiologie
Celsiusstr. 1
D-28359 Bremen
|
Raum: |
2222 |
|
Telefon: |